|  |
Die vorgestellten Häufigkeitsmaße kann man anhand der angegebenen Formeln mit einem geeigneten Desktop- oder Taschenrechner berechnen. Auch die Software R kann man als Taschenrechner verwenden, aber mit R kann man auch viel mehr, wie wir sehen werden.
Anders als DWDS ist R keine Internet-Anwendung, sondern man muss es herunterladen und auf einen Computer installieren, was i.d.R. problemlos ist. (R ist sogenannte „freie“ Software, d.h. nicht nur kostenlos, sondern man kann auch den gesamten Quellcode ansehen und ändern und die Änderungen auch anderen Nutzern zur Verfügung stellen.)
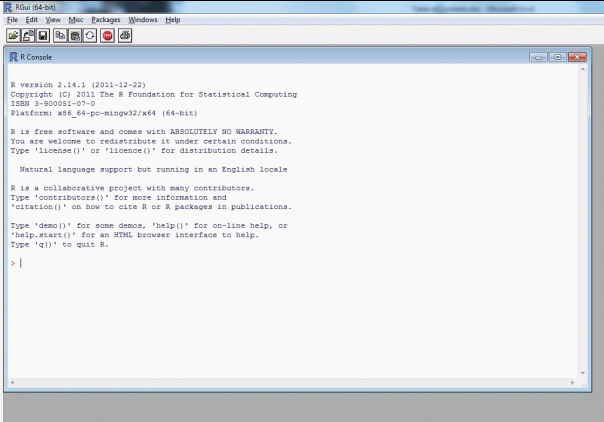
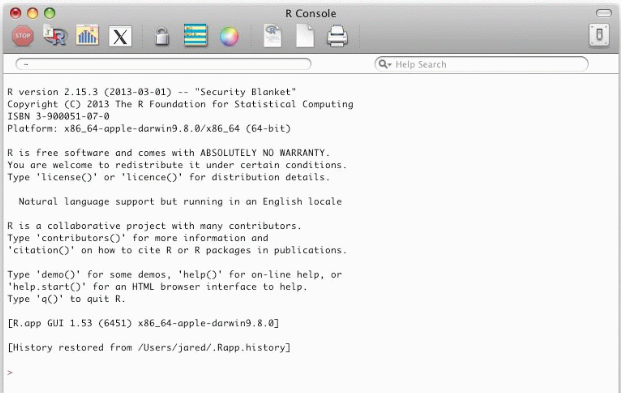
Die eingebaute Benutzerschnittstelle von R ist eine einfache Kommandozeile, in die man Daten und Befehle eingibt (indem man sie eintippt oder auch per Copy-&-Paste). Klickt man unter MS-Windows oder macOS auf das R-Icon am Desktop oder ruft R z.B. vom Start-Menü auf, öffnet sich ein Fenster, das die R-Konsole (die Kommandozeile) enthält:

|  |
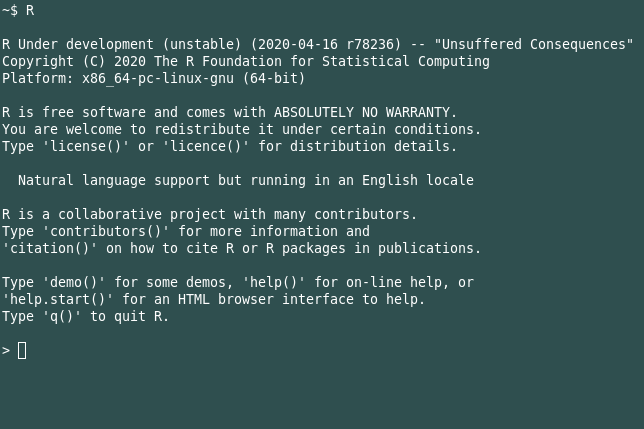
Unter Linux gibt man einfach den Programmnamen "R" in die Shell (die Linux-Kommandozeile) ein:
N.B.: Wenn die Sprache in Ihrem Betriebssystem auf Deutsch eingestellt ist, sollten die Meldungen, die R ausgibt, auch auf deutsch sein. Dennoch müssen Sie numerische Eingaben in R nach der englischen Schreibweise machen, siehe gleich unten.
Um R als Taschenrechner zu benutzen, gibt man Zahlen und arithmetische Zeichen direkt in die R-Kommandozeile ein und drückt die Eingabetaste, um die Berechnungen auszuführen. Die Ein- und Ausgaben für die Berechnungen der relativen Häufigkeit des Lemmas ‚Hund‘ im DWDS-Kernkorpus als Dezimalzahl, in Prozent bzw. in pMW sehen z.B. so aus:
> 7279 / 121397601
[1] 5.996e-05
> 7279 / 121397601 * 100
[1] 0.005996
> 7279 / 121397601 * 1e6
[1] 59.96
Anmerkungen:
>‘ gehört nicht zur Eingabe
sondern stellt die Eingabeaufforderung (den R-Kommandozeilenprompt)
dar, um zwischen aufeinanderfolgenden Eingaben zu unterscheiden.
> 7279 / 121397601 *
+ 1000000
[1] 59.96
[n]‘, die bedeutet, dass der folgende
Wert das n-te Element der Ausgabe ist. In den obigen
Berechnungen besteht die Ausgabe aus einem einzigen Element, also ist
die Angabe ‚[1]‘ hier überflüssig, aber wir
werden Berechnungen sehen, bei denen die Ausgabe viele Elemente über
mehrere Zeilen enthält und dann sind solche Angaben hilfreich.
> 181 / (181 + 736) * 100
[1] 19.73828
> 181 / 181 + 736 * 100
[1] 73601
Für dieses Seminar müssen Sie zwar nicht mit R programmieren, aber für eine korrekte und effiziente Verwendung von R ist es dennoch sinnvoll, gewisse Eigenschaften von R als Programmiersprache kennenzulernen.
Oft will man bestimmte Eingaben oder auch Ergebnisse bestimmter Berechnungen wiederholt verwenden. Das ist z.B. der Fall bei der Berechnung des Differenzenkoeffizienten, wo die erwartete Häufigkeit zuerst berechnet und das Ergebnis dann zweimal verwendet wird. Wiederverwendbarkeit ist eines der Hauptmerkmale einer Programmiersprache und das R-System verfügt auch über eine Programmiersprache (die auch R heißt). In Programmiersprachen wird Wiederverwendbarkeit durch sogenannte Variablen unterstützt.
Hund‘, ‚hund‘
und ‚HUND‘ stellen unterschiedliche
Variablen dar.hund.kk.fa für die absolute Häufigkeit von
‚Hund‘ im DWDS-Kernkorpus. (Leerzeichen sind in
Variablennamen unzulässig und ‚-‘ ist als Trennzeichen
nicht empfehlenswert, weil es Gefahr läuft, von R als Subtraktion
verarbeitet zu werden.)<-‘ von rechts
nach links:> hund.kk.fa <- 7279<‘ und ‚-‘
ohne Leerzeichen dazwischen. (Man kann allerdings auch
‚->‘ und ‚=‘
als Zuweisungsoperatoren verwenden, wobei das Gleichheitszeichen
bevorzugt nur für Zuweisungen an Funktionsparametern (siehe unten)
verwendet wird, und mit ‚->‘ steht der
zugewiesene Wert auf der linken Seite: 7279 ->
hund.kk.fa .)
> hund.kk.fa
[1] 7279
> (katze.kk.fa <- 2359)
[1] 2359
Die folgenden Eingaben in der R-Kommandozeile zeigen, wie man den Differenzenkoeffizienten des Lemmas ‚Hund‘ im DWDS-Kernkorpus für die Dekade 1960-1969 als Teilkorpus anhand der vorher angegebenen Formeln unter Verwendung von Variablen in R berechnen kann:
> kk.groesse <- 121397601
> kk.1960.groesse <- 10587420
> hund.fb <- 474
> (hund.fe <- hund.kk.fa * kk.1960.groesse / kk.groesse)
[1] 634.8217
> (hund.fb - hund.fe) / (hund.fb + hund.fe)
[1] -0.1450384
Für die Berechnung von Häufigkeitsklassen in R nach der angegeben Formel brauchen wir einen weiteren Bestandteil von Programmiersprachen, Funktionen:
Hier ist die Berechnung in R der Häufigkeitsklasse des Lemmas ‚Hund‘ im DWDS-Kernkorpus; sie enthält zwei Funktionsaufrufe, die ineinander verschachtelt sind:
> kk.ref.fa <- 11113606
> round(log2(kk.ref.fa / hund.kk.fa))
[1] 11
Anmerkungen:
log2() nimmt eine Zahl als
Argument – hier das Ergebnis der Division der absoluten
Häufigkeit des Referenzlemmas ‚der‘ durch die absolute
Häufigkeit des Lemmas ‚Hund‘ – und berechnet den
Logarithmus dieser Zahl zur Basis 2.round() nimmt eine Zahl als
Argument – hier den Rückgabewert
des log2()-Funktionsaufrufs – und gibt die nächste
ganze Zahl zurück (lässt also die Nachkommazahlen weg). Damit werden
die Häufigkeitsklassen immer durch ganze Zahlen dargestellt.Außer sehr vielen eingebauten Funktionen (von denen wir im Laufe dieses Seminars noch einige kennenlernen werden) gibt es in R, wie in jeder Programmiersprache, auch die Möglichkeit eigene Funktionen zu definieren.
R hat z.B. keine eingebauten Funktionen für die Berechnung der vorher vorgestellten Häufigkeitsmaße. Zwar könnten wir diese wie oben durch Folgen von Eingaben in der Kommandozeile berechnen, aber für wiederholte Berechnungen mit verschiedenen Werten wäre es viel praktischer, sie einfach durch Funktionsaufrufe zu machen. Daher haben wir solche Funktionen definiert, und dazu noch eine Funktion zur Erstellung einer Tabelle von Häufigkeitsklassen; ihre Namen und Argumente lauten wie folgt:
Diese Funktionen sind so definiert, dass sie beim Aufruf die oben gezeigten Berechnungsschritte „unter der Haube“ durchführen und als Ergebnis das entsprechende Häufigkeitsmaß ausgeben.
Ein wesentlicher Unterschied zwischen eingebauten und selbst-definierten Funktionen ist, dass die eingebauten Funktionen schon beim Start der R-Sitzung zur Verfügung stehen und sofort aufgerufen werden können, während selbst-definierte Funktionen R zuerst bekannt gemacht werden müssen, bevor sie verwendet werden können. Eine Möglichkeit der Bekanntmachung ist, die Funktion gleich in der R-Kommandozeile zu definieren (z.B. könnten Sie die unten angegebenen Definitionen per Copy-&-Paste in R eingeben).
Aber insbesondere wenn man viele selbst-definierten Funktionen benutzen
will, ist es praktischer, die Definitionen in einer Datei zu speichern
und diese durch Aufruf der eingebauten R-Funktion source()
mit dem Namen der Datei als Argument in R laden. Der Dateiname kann
auch die URL (Adresse) einer Seite im Internet sein, dann wird diese in
R heruntergeladen. Eine solche Seite mit eigens für dieses Seminar
definierten R-Funktionen, darunter die für die vorgestellten
Häufigkeitsmaße, habe ich eingerichtet. Um also diese Funktionen in R
zu laden, geben Sie folgende Anweisung in R ein:
> source("http://homepage.rub.de/Stephen.Berman/Korpuslinguistik/Funktionen.R", encoding="UTF-8")
Anmerkung:
encoding="UTF-8" sorgt für eine korrekte
Darstellung der Datei in R unter dem Betriebssystem MS-Windows; ohne
diese Angabe können Zeichen wie z.B. Umlaute falsch dargestellt
werden in R.Nach dieser Eingabe stehen in der aktuellen R-Sitzung die
Funktionen freq.rel(), freq.prop(), diff.koeff()
und freq.klasse() zur Verfügung. Damit können die obigen
Berechnungen der Häufigkeitsmaße noch einmal durchgeführt werden, jetzt
einfach als Funktionsaufrufe. Dabei gibt es mehrere völlig äquivalente
Formen des Aufrufs, für freq.rel() z.B. folgende:
> freq.rel(freq.absolut = 7279, korp.groesse = 121397601)
[1] 59.96
> freq.rel(freq.absolut = hund.kk.fa, korp.groesse = kk.groesse)
[1] 59.96
> freq.rel(7279, 121397601)
[1] 59.96
> freq.rel(hund.kk.fa, kk.groesse)
[1] 59.96
Die ersten zwei Formen enthalten explizite Zuweisungen von Werten an die Argumente (dafür wird in R das Gleichheitszeichen statt ‚<-‘ verwendet); in der ersten Form wird der Wert direkt als Zahl übergeben, in der zweiten Form mittels einer Variable, der der Zahlenwert vor dem Funktionsaufruf zugewiesen worden ist. Bei Funktionsaufrufen mit expliziten Argumentzuweisungen ist die Reihenfolge der Argumente unerheblich:
> freq.rel(korp.groesse = 121397601, freq.absolut = 7279)
[1] 59.96
In der dritten und vierten Form des Aufrufs werden nur die Argumentwerte angegeben (in der dritten direkt als Zahl, in der vierten mittels einer Variable). Bei solchen Aufrufen muss die Reihenfolge der Werte mit der Reihenfolge der Argumente in der Funktionsdefinition genau übereinstimmen, sonst kann das Ergebnis inkorrekt sein:
> freq.rel(121397601, 7279)
[1] 16677785547
Daher sind Funktionsaufrufe mit expliziten Argumentzuweisungen sicherer – und meist auch lesbarer.
Wie oben angegeben nimmt die Funktion freq.rel() auch noch
ein drittes Argument namens ‚type‘, das bestimmt, ob das
Ergebnis in pMW, Prozent oder als Dezimalzahl ausgegeben wird. Anders
als die Argumente ‚freq.absolut‘ und
‚korp.groesse‘ hat ‚type‘
einen voreingestellten Wert, nämlich "pMW". Argumente mit einem
voreingestellten Wert dürfen beim Funktionsaufruf weggelassen werden,
dann wird dieser Wert automatisch übergeben (deswegen sind die Ausgaben
der oben gezeigten Aufrufe von freq.rel() alle in pMW).
Möchte man einen nicht voreingestellten Wert des Arguments verwenden,
muss dieser explizit angegeben werden (N.B.: Der Wert des
‚type‘-Arguments muss in Anführungsstrichen stehen):
> freq.rel(freq.absolut = 7279, korp.groesse = 121397601, type = "dezimal")
[1] 5.996e-05
> freq.rel(freq.absolut = 7279, korp.groesse = 121397601, type = "prozent")
[1] 0.005996
> freq.rel(freq.absolut = 7279, korp.groesse = 121397601, type = "pMW")
[1] 59.96
Hier sind noch Aufrufe der
Funktionen diff.koeff(), freq.klasse()
und freq.klasse.tab() mit expliziten Argumentzuweisungen;
diese stellen keine Besonderheiten dar:
> diff.koeff(freq.absolut = 7279, korp.groesse = 121397601,
freq.beobachtet = 474, teilkorp.groesse = 10587420)
[1] -0.1450384
> freq.klasse(freq.absolut.w = 7279, freq.absolut.ref = 11113606)
[1] 11
> freq.klasse.tab(freq.absolut.ref = 11113606)
hk min max
[1,] 0 7858507 11113606
[2,] 1 3929254 7858506
[3,] 2 1964627 3929253
[4,] 3 982314 1964626
[5,] 4 491157 982313
[6,] 5 245579 491156
[7,] 6 122790 245578
[8,] 7 61395 122789
[9,] 8 30698 61394
[10,] 9 15349 30697
[11,] 10 7675 15348
[12,] 11 3838 7674
[13,] 12 1919 3837
[14,] 13 960 1918
[15,] 14 480 959
[16,] 15 240 479
[17,] 16 120 239
[18,] 17 60 119
[19,] 18 30 59
[20,] 19 15 29
[21,] 20 8 14
[22,] 21 4 7
[23,] 22 2 3
[24,] 23 1 1
Anmerkung:
Bei der Funktion freq.prop() gibt es aber sehr wohl eine
Besonderheit: Nach der Formel
für proportionale Häufigkeiten sollen die Verhältnisse von mindestens
zwei aber im Prinzip beliebig vielen Suchergebnissen berechnet werden
können. In R kann dieser Möglichkeit durch einen speziellen Datentyp
namens Vektor Rechnung getragen werden: Ein Vektor ist
eine Folge von Daten derselben Art (z.B. Zahlen wie 1, 2 oder
Zeichenketten wie "abc", "def"). Das Argument
‚freqs.absolut‘ von freq.prop() nimmt als Wert
also einen Vektor von (zwei oder mehr) Zahlen (N.B.: Im Namen des
Arguments steht ‚freqs‘ und nicht ‚freq‘, um
anzudeuten, dass der Vektor mindestens zwei Zahlen enthalten soll).
Für die Eingabe von Vektoren gibt es in R verschiedene Möglichkeiten,
von denen die eingebaute Funktion c() wohl am allgemeinsten
und am nützlichsten ist (der Name ‚c‘ steht für das
englische Wort concatenate, etwa verketten, verknüpfen,
aneinanderhängen). Diese Funktion nimmt als Argumente beliebig viele
durch Kommata getrennte Elemente desselben Datentyps. Hier sind also
mögliche Aufrufe der Funktion freq.prop():
> freq.prop(freqs.absolut = c(181, 736))
[1] 19.74 80.26
> freq.prop(freqs.absolut = c(181, 736, 234))
[1] 15.73 63.94 20.33
> freq.prop(freqs.absolut = c(181, 736, 234, 589))
[1] 10.40 42.30 13.45 33.85
Anmerkung:
freqs.absolut ist.
Also ist z.B. im ersten Aufruf 19.74 die proportionale Häufigkeit von
181 und 80.26 die proportionale Häufigkeit von 736.
> gesendet.kk.fa <- 181
> gesandt.kk.fa <- 736
> freq.prop(freqs.absolut = c(gesendet.kk.fa, gesandt.kk.fa))
[1] 19.74 80.26
Um Funktionen zu definieren, verwendet man die eingebaute
R-Funktion function():
return() sein.Die Definitionen der Funktionen für Differenzenkoeffizienten und Häufigkeitsklassen sind direkte Übertragungen der Anweisungen in den obigen Beispielen:
diff.koeff <- function(freq.absolut, korp.groesse, freq.beobachtet, teilkorp.groesse) {
freq.erwartet <- freq.absolut * teilkorp.groesse / korp.groesse
(freq.beobachtet - freq.erwartet) / (freq.beobachtet + freq.erwartet)
}
freq.klasse <- function(freq.absolut.w, freq.absolut.ref) round(log2(freq.absolut.ref / freq.absolut.w))
Die Definitionen der Funktionen für relative und proportionale Häufigkeiten, sowie für Tabellen von Häufigkeitsklassen, sind komplizierter, weil sie bestimmte Programmiertechniken oder weitere eingebaute R-Funktionen verwenden:
freq.rel <- function(freq.absolut, korp.groesse, type="pMW") {
fr <- freq.absolut / korp.groesse
if (type == "dezimal")
freq <- fr
else if (type == "prozent")
freq <- fr * 100
else freq <- fr * 1e6 # Darstellung in pMW
return(freq)
}
Anmerkungen:
if … else … formuliert wird. Der
Wert des Parameters type bedingt die Ausgabe: Ist
dieser Wert "dezimal" (n.b.: die Anführungsstriche müssen mit
angegeben werden), wird die relative Häufigkeit als Dezimalzahl
ausgegeben, ist der Wert "prozent", wird die relative Häufigkeit
in Prozent ausgegeben. Die Ausgabe in pMW ist durch die
Funktionsdefinition voreingestellt und muss daher nicht explizit
angegeben werden beim Aufruf (aber man kann das
mit type="pMW" auch tun).#‘ vor
(aber nicht innerhalb von Anführungsstrichen), dann gelten alle
folgenden Zeichen in derselben Zeile als Kommentar, der von R
ignoriert, also nicht verarbeitet, wird. Kommentare werden zur
Erläuterung des Programmcodes verwendet.freq.prop <- function(freqs.absolut) {
summe <- sum(freqs.absolut)
fp <- NULL
for (i in 1:length(freqs.absolut))
fp[i] <- freqs.absolut[i] / summe * 100
round(fp, 2)
}
Anmerkungen:
sum() nimmt einen Vektor
von Zahlen als Argument und gibt die Summe der Zahlen zurück, also
bewirkt z.B. die Anweisung sum(c(1, 2, 3, 4, 5))
dasselbe wie 1 + 2 + 3 + 4 + 5.freqs.absolut durch diese Summe geteilt und die
Ergebisse jeweils dem entsprechenden Element des
Ausgabe-Vektors fp zugewiesen.
:‘ bildet
einen Vektor von ganzen Zahlen, hier von 1 bis zur Stellenwert
des letzten Elements von freqs.absolut (diesen
Stellenwert berechnet die eingebaute
Funktion length()).i wird der jeweilige Stellenwert
zugewiesen und fp[i]
bzw. freqs.absolut[i] stellen das i-te Element
des entsprechenden Vektors dar (wir werden diese Notation und
ihre Bedeutung später im Zusammenhang mit Datensätzen
detailliert besprechen).fp muss durch Zuweisung
initialisiert, d.h. R bekannt gemacht werden; das
R-Schlüsselwort NULL dient als leeren Wert für die
Initialisierung des Vektors.freqs.absolut. Durch round(fp,
2) werden die proportionalen Häufigkeiten auf zwei
Nachkommazahlen gerundet. Diese Anweisung zeigt,
dass round() nicht nur eine Zahl sondern auch einen
Vektor von beliebig vielen Zahlen als Argument nehmen kann.
(Eigentlich behandelt R in diesem Zusammenhang eine Zahl als einen
Vektor, der nur diese Zahl als Element enthält und somit kann man
behaupten, dass das erste Argument von round()
einfach ein Vektor von einer oder mehr Zahlen ist.)freq.klasse.tab <- function(freq.absolut.ref) {
n <- freq.klasse(1, freq.absolut.ref.ref)
max <- floor(freq.absolut.ref / 2^seq(0.5, n))
min <- max + 1
max <- c(freq.absolut.ref, max)
min <- c(min, 1)
hk <- 0:n
cbind(hk, min, max)
}
Anmerkungen:
n speichert die Zahl der höchsten
Häufigkeitsklasse, wie sie durch Aufruf der oben definierten
Funktion freq.klasse() berechnet wird, indem dieser
Funktion die Wortfrequenz 1 für die höchste Häufigkeitsklasse (die
Klasse der Hapax legomena) übergeben wird.max und min in dieser
Definition speichern die Grenzwerte der Häufigkeitsklassen
und hk speichert den Vektor der Zahlen, die die
Häufigkeitsklassen bezeichnen.floor() nimmt einen Vektor
von Zahlen als das Argument und gibt einen Vektor von ganzen
Zahlen zurück, die jeweils kleiner als die entsprechende Zahl des
Argument-Vektors sind (z.B. floor(2.7) ergibt 2).^‘ ist die Umkehrfunktion des
Logarithmus (d.h., ist y gleich 2^x, dann ist x
gleich log2(y)). Auch diese Funktion nimmt einen
Vektor von Zahlen als Argument.seq() bildet einen Vektor
von Zahlen, der beginnt mit dem Wert des ersten Arguments und
endet mit dem Wert des zweiten Arguments; das Intervall zwischen
den Werten ist voreingestellt als 1, kann durch Übergabe eines
dritten Argumentwerts geändert werden.cbind() bildet eine
Tabelle, dessen Spalten aus den Argumentwerten
von cbind() (die Vektoren sind) bestehen. (Der Name
‚cbind‘ steht für „bind columns“, also
Spalten verbinden.)(Zur Information: Die Operatoren ‚^‘ und
‚:‘ sowie die arithmetischen Operatoren sind
eigentlich Funktionen in R, die jedoch normalerweise in der vertrauteren
mathematischen Schreibweise als binäre Operatoren zwischen ihren
Argumenten verwendet werden. Aber auch die Verwendung in Form eines
Funktionsaufrufs ist möglich, dann muss allerdings das Zeichen, das
eigentlich der Name der Funktion ist, in Anführungsstriche gesetzt
werden: z.B. "*"(2, 3), "/"(2, 3), "+"(2, 3), "-"(2, 3), "^"(2, 3),
":"(2, 3). Selbst die Variablenzuweisung kann in Form eines
Funktionsaufrufs erfolgen: "<-"(mein.var, 123). Dennoch sollten Sie
der Lesbarkeit halber i.d.R. nur die Schreibweise als binäre Operatoren
verwenden.)
Wenn Sie die Darstellung von Dezimalzahlen mit Nullen nach dem Punkt
der Exponentialdarstellung vorziehen, können Sie Letztere mit der
eingebauten R-Funktion options() und der eingebauten
R-Variable scipen, der Sie einen hohen Zahlenwert zuweisen,
unterbinden und anschließend wiederherstellen:
> freq.rel(7279, 121397601, type="dezimal")
[1] 5.996e-05
> scipen.orig <- options(scipen = 999)
> freq.rel(7279, 121397601, type="dezimal")
[1] 0.00005996
> options(scipen = scipen.orig)
> freq.rel(7279, 121397601, type="dezimal")
[1] 5.996e-05
Anmerkung:
scipen.orig <- options(scipen = 999)
speichert den ursprünglichen Wert der eingebauten
Variable scipen in der Variable scipen.org
und gleichzeitig weist scipen den neuen Wert 999 zu. Die
Anweisung options(scipen = scipen.orig) stellt den
ursprünglichen Wert wieder her.Sie können die laufende R-Sitzung durch Aufruf der eingebauten
R-Funktion q() (ohne Argumente) in der Kommandozeile
beenden; unter MS-Windows oder macOS können Sie die Sitzung auch über
das File-Menü beenden.
Bevor die Sitzung beendet wird, fragt R, ob die Daten der Sitzung – die Variablen und Funktionen, die Sie definiert haben – gespeichert werden sollen. Antworten Sie mit ja, stehen dieselben Daten zu Beginn der nächsten Sitzung wieder zur Verfügung, Sie müssen sie also nicht noch einmal in R eingeben – vorausgesetzt, Sie starten R im selben Ordner (Verzeichnis) Ihres Rechners, in dem Sie die letzte Sitzung beendet haben. (Die Datei, in der die Daten gespeichert sind, hat den Namen „.RData“; diese ist keine Text-Datei, kann aber in R geladen werden.)
Variablenzuweisungen, Funktionsaufrufe usw., die Sie in der laufenden Sitzung gemacht haben, können Sie mit Hilfe der Pfeiltasten (nach oben und nach unten) wieder in die Eingabeaufforderung bringen, ohne sie zu erneut eintippen zu müssen. Das gilt auch für solche Eingaben aus früheren Sitzungen bei normalen R-Installationen. (Die Datei, in der alle Eingaben gespeichert sind, hat den Namen „.Rhistory“; diese ist eine Text-Datei, Sie können sie also mit einem Texteditor lesen und damit alle Eingaben, die Sie gemacht haben, nachvollziehen.)
Wenn Sie während oder vor Beenden der laufenden Sitzung bestimmte Daten
löschen wollen, können Sie das durch Aufruf der eingebauten
R-Funktion rm(), wobei die Daten, d.h. die Namen der
Variablen und Funktionen, als durch Kommata getrennte Argumente
von rm() angegeben werden müssen (z.B. rm(hund.fa,
katze.fa, maus.fa)). Vorher können Sie durch Aufruf der
eingebauten R-Funktion ls() (ohne Argumente) eine
Auflistung aller verwendeten Variablen und Funktionen erhalten. Sie
können alle von Ihnen verwendeten Daten mit folgender Eingabe auf einmal
löschen: rm(list=ls()).