Hypothesentests haben im Allgemeinen folgendes Ablaufschema.
In diesem Ablaufschema wird nur ein möglicher Fehler, den man bei der Entscheidung über die statistische Signifikanz der Teststatistik begehen kann, angesprochen: die Ablehnung der Nullhypothese, obwohl sie in Wirklichkeit (d.h. in der Grundgesamtheit) gilt (aber eben nicht in der Stichprobe). Dieser Fehler heißt der Fehler 1. Art (oder α-Fehler). Aber es gibt auch einen anderen möglichen Fehler, den Fehler 2. Art (oder β-Fehler): die Beibehaltung der Nullhypothese, obwohl in der Grundgesamtheit die Alternativhypothese gilt (aber eben nicht in der Stichprobe).
Es gibt viele spezifische Hypothesentests für statistische Untersuchungen, die sich in der Anzahl und Art (Skalenniveau) der untersuchten Variablen oder in der Anzahl der untersuchten Stichproben unterscheiden, die aber alle nach dem oben geschilderten Ablaufschema durchgeführt werden. Und genauso wie es eingebaute R-Funktionen für viele Wahrscheinlichkeitsverteilungen gibt, gibt es auch für viele Hypothesentests eingebaute R-Funktionen. Diese nehmen als Argumente die Stichprobenwerte sowie die Parameterwerte der zugrundeliegenden Wahrscheinlichkeitsverteilung, und berechnen damit den p-Wert, von dem man anhand des gewählten Signifikanzniveaus auf die statistische Signifikanz des Stichprobenwerts schließt; oft berechnen sie auch Konfidenzintervalle.
Weiter unten werden drei R-Funktionen vorgestellt, die für Hypothesentests mit binomialverteilten Zufallsvariablen und generell für Häufigkeiten von nominalen Variablen geeignet sind. Diese Tests spielen in korpuslinguistischen statistischen Recherchen eine wichtige Rolle, nicht zuletzt aufgrund der Annahme, dass Wortfrequenzen in Korpora durch binomialverteilte Zufallsvariablen statistisch modelliert werden können.
Es ist aber auch sinnvoll, etwas davon zu verstehen, wie die p-Werte zustandekommen und warum sie einen Rückschluss auf statistische Signifikanz ermöglichen. Das zeigen wir im Detail in den zwei folgenden Abschnitten „Berechnung des p-Werts“. Diese Erläuterungen sollen dem Verständnis dienen; aber in normalen statistischen Untersuchungen mit R (und auch in den Übungsaufgaben) verwendet man nur die spezifischen Funktionen für Hypothesentests, wie die drei, die weiter unten vorgestellt werden.
Ein Ein-Stichprobentest eignet sich, um z.B. ein früheres Forschungsergebnis zu einer Eigenschaft der Grundgesamtheit anhand einer neuen Beobachtung aus einer Stichprobe zu überprüfen. Im Folgenden geben wir ein freilich unrealistisches Beispiel, um die wesentlichen Schritte der Methode möglichst genau aber auch verständlich zeigen zu können.
source-Aufruf
durchführen, um freq.rel() in R zu laden):
> kk.gr <- 121397601
> hund.kk.fa <- 4395
> (hund.kk.fr <- freq.rel(hund.kk.fa, kk.gr, "dezimal"))
[1] 3.620335e-05
Gemäß der Annahme, dass Wortfrequenzen binomialverteilte Zufallsvariablen sind, ergibt unsere Nullhypothese eine Stichprobenverteilung basierend auf folgende Zufallsvariable:
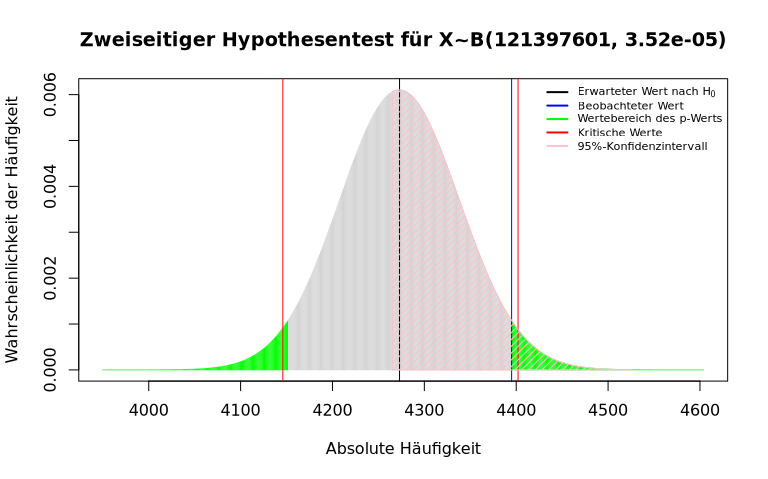
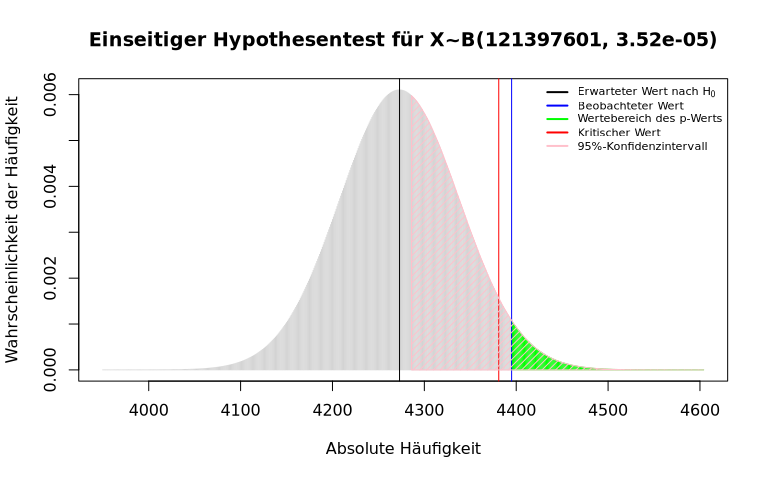
X ∼ B(121397601, 3.52e-05)
pbinom(),
die die Verteilungsfunktion der Binomialverteilung darstellt. Da die
relative Häufigkeit des beobachteten Werts (3.620335e-05) größer als
die relative Häufigkeit nach der Nullhypothese (3.52e-05) ist,
berechnen wir zunächst die Summe der Wahrscheinlichkeit des
beobachteten Werts und der Wahrscheinlichkeiten aller größeren Werte,
also die Summe der Werte am oberen Ende der Verteilung:
> (hund.kk.pwo <- pbinom(q = hund.kk.fa - 1, size = kk.gr, prob = 3.52e-05, lower.tail = FALSE))
[1] 0.0321884
lower.tail=FALSE berücksichtigt
pbinom() nur Werte, die kleiner oder gleich dem Wert
des ersten Arguments sind, aber mit lower.tail=FALSE
werden nur die übrigen Werte berücksichtigt – also nur die,
die größer als der Wert des ersten Arguments sind. Daher muss von
diesem 1 abgezogen werden, damit auch die Wahrscheinlichkeit der
beobachteten absoluten Häufigkeit selbst mit berechnet wird (es
handelt sich um eine diskrete Verteilung, deren Werte
ausschließlich ganzen Zahlen sind).Weil wir einen zweiseitigen Test durchführen, müssen wir auch Werte
am unteren Ende der Verteilung berücksichtigen. Wäre die
Stichprobenverteilung symmetrisch, würde die Summe dieser
Wahrscheinlichkeiten dieselbe wie am oberen Ende sein; aber
Binomialverteilungen sind nur dann symmetrisch, wenn der Parameter
der Wahrscheinlichkeit p = 0,5 ist, was hier nicht der Fall
ist. Es liegt aber nahe, alle Werte zu berücksichtigen, die
mindestens so viel kleiner sind als der erwartete Wert gemäß der
Nullhypothese, als der beobachtete Wert größer als dieser ist. Wir
berechnen also die Differenz zwischen dem beobachteten und dem
erwarteten Wert und ziehen diese vom erwarteten Wert ab. Dann
berechnen wir mit pbinom() die Summe der
Wahrscheinlichkeiten dieses Werts und aller kleineren Werte (hier
hat das Argument lower.tail den Wert TRUE, und weil
dieser Wert voreingestellt ist, können wir das Argument weglassen).
Hier sind die R-Eingaben und -Ausgaben dieser Berechnungen:
# Der erwartete Wert gemäß der Nullhypothese:
> (hund.kk.fe <- round(3.52e-05 * kk.gr))
[1] 4273
round(), um eine ganze Zahl zu erhalten,
weil es sich ja um eine diskrete Verteilung handelt.
# Der Wert, der kleiner als der erwartete Wert ist und denselben Abstand zu diesem hat als der ermittelte Wert:
> (hund.kk.fa2 <- hund.kk.fe - (hund.kk.fa - hund.kk.fe))
[1] 4151
# Die Summe der Wahrscheinlichkeiten der kleineren Werte:
> (hund.kk.pwu <- pbinom(q = hund.kk.fa2, size = kk.gr, prob = 3.52e-05))
[1] 0.03087651
Der gesuchte p-Wert ist dann die Summe dieser summierten größeren und kleineren Wahrscheinlichkeiten:
> hund.kk.pwo + hund.kk.pwu
[1] 0.06306491
Vergleichen wir dieses Ergebnis mit dem Ergebnis des entsprechenden einseitigen Tests:
hund.kk.pwo ≈ 0,032Das Fazit dieses Beispiels: Hätten wir Grund zur Annahme des einseitigen Tests, würde das Ergebnis der Stichprobe als Evidenz für eine Änderung unseres Wissensstands zählen, aber ohne Grund zu dieser Annahme können wir den aktuellen Wissensstand nicht in Frage stellen.
Hier zum Abschluss dieses Beispiels sind grafische Darstellungen der zwei durchgeführten Hypothesentests, die auch entsprechende Konfidenzintervalle zeigen:


Anmerkungen:
qbinom() berechnet werden, für das aktuelle Beispiel
also wie folgt (die erste Berechnung für den einseitigen Test, die
zweite und dritte für den zweiseitigen):
> qbinom(p = .05, size = 121397601, prob = 3.52e-05, lower.tail = FALSE)
[1] 4381
> qbinom(p = .05/2, size = 121397601, prob = 3.52e-05, lower.tail = FALSE)
[1] 4402
> qbinom(p = .05/2, size = 121397601, prob = 3.52e-05)
[1] 4146
Normalerweise würde man den p-Wert eines Ein-Stichprobentests für eine
binomialverteilte Zufallsvariable nicht wie oben manuell in R berechnen
sondern einfach mit einem Aufruf der
Funktion binom.test():
binom.test() nimmt als obligatorische Argumente:
alternative mit einem der Werte "greater" oder
"less" für einen einseitigen Hypothesentest, oder dem Wert
"two.sided" für einen zweiseitigen Test (letzterer ist der
voreingestellte Wert).conf.level mit einer Zahl als Wert des
Konfidenzniveaus für die Berechnung des Konfidenzintervalls (der
voreingestellte Wert ist 0.95).Die Ausgabe dieser Funktionen ist ein kleiner Bericht über den Hypothesentest, der u.a. die Eingabedaten des Tests, den p-Wert und das Konfidenzintervall enthält. Hier sind z.B. die Ein- und Ausgaben für die obigen zwei- bzw. einseitigen Ein-Stichprobentests:
> binom.test(hund.kk.fa, kk.gr, 3.52e-05)
Exact binomial test
data: hund.kk.fa and kk.gr
number of successes = 4395, number of trials = 121397601, p-value = 0.06306
alternative hypothesis: true probability of success is not equal to 3.52e-05
95 percent confidence interval:
3.514086e-05 3.728980e-05
sample estimates:
probability of success
3.620335e-05
> binom.test(hund.kk.fa, kk.gr, 3.52e-05, alternative = "greater")
Exact binomial test
data: hund.kk.fa and kk.gr
number of successes = 4395, number of trials = 121397601, p-value = 0.03219
alternative hypothesis: true probability of success is greater than 3.52e-05
95 percent confidence interval:
3.530983e-05 1.000000e+00
sample estimates:
probability of success
3.620335e-05
Die p-Werte hier sind identisch mit den oben berechneten p-Werten (aber
R rundet sie hier auf fünf Nachkommastellen ab bzw. auf), was darauf
zurückzuführen ist, dass der Funktion binom.test() die
Wahrscheinlichkeitsfunktion
der Binomialverteilung zugrundeliegt. Damit sind die berechneten
Wahrscheinlichkeiten zwar exakt, allerdings gibt es dazu eine
Kehrseite:
binom.test()
kann sehr hoch sein und mehr Speicher erfordern, als der Rechner zur
Verfügung stellt, was zum Einfrieren oder Abbruch von R führen kann.
Das kann z.B. passieren, wenn die Anzahl der Stufen (was für uns der
Korpusgröße entspricht) sehr groß ist und sich die beobachtete
relative Häufigkeit nur leicht von der Wahrscheinlichkeit des Erfolgs
abweicht.Falls man auf dieses Problem stößt, gibt es eine Alternative: die
R-Funktion prop.test(), die viel weniger
Rechenaufwand erfordert. Sie wird mit den gleichen Argumenten
wie binom.test() aufgerufen und gibt einen ähnlichen
Bericht über den Hypothesentest aus:
> prop.test(hund.kk.fa, kk.gr, 3.52e-05)
1-sample proportions test with continuity correction
data: hund.kk.fa out of kk.gr, null probability 3.52e-05
X-squared = 3.4436, df = 1, p-value = 0.0635
alternative hypothesis: true p is not equal to 3.52e-05
95 percent confidence interval:
3.514469e-05 3.729378e-05
sample estimates:
p
3.620335e-05
> prop.test(hund.kk.fa, kk.gr, 3.52e-05, alternative = "greater")
1-sample proportions test with continuity correction
data: hund.kk.fa out of kk.gr, null probability 3.52e-05
X-squared = 3.4436, df = 1, p-value = 0.03175
alternative hypothesis: true p is greater than 3.52e-05
95 percent confidence interval:
3.531212e-05 1.000000e+00
sample estimates:
p
3.620335e-05
Die p-Werte hier sind zwar keine exakten Werte wie
beim binom.test() (mit prop.test() ist der
p-Wert beim zweiseitigen Test etwas höher und beim einseitigen Test
etwas kleiner), aber die Annäherung ist bei großen Stichproben so gut,
dass die Schlussfolgerungen bezüglich statistischer Signifikanz
unverändert bleiben.
prop.test() auf der Chi-Quadrat-Verteilung
basiert und verwendet
die Normal-Approximation
der Binomialverteilung. Die Normalverteilung ist ja
eine symmetrische Verteilung, folglich ist
mit prop.test() der p-Wert beim zweiseitigen Test genau
doppelt so groß wie beim einseitigen Test, was
mit binom.test() nicht der Fall ist.Auch die mit binom.test() und prop.test()
berechneten Konfidenzintervalle unterscheiden sich leicht voneinander,
und auch von denen, die wir auf der vorigen Seite
mit konfint.fr() und konfint.fr.diff()
berechnet haben. Die von binom.test()
und prop.test() verwendeten Berechnungen ergeben
insbesondere bei kleinen Stichprobengrößen bessere Ergebnisse, sind
dafür aber mathematisch komplizierter. Für die in der Korpuslinguistik
üblichen Stichprobengrößen sind diese Unterschiede
i.d.R. unerheblich.
Anstatt der Ausgabe als Bericht kann man den Rückgabewert
von binom.test() und prop.test(), der eine
Liste der Daten und Ergebnisse des Hypothesentests ist, einer Variable
zuweisen und dann mit Hilfe des $-Operators auf die Elemente der Liste
zugreifen. Z.B. werden mit folgenden Eingaben nur die p-Werte der
obigen zwei- und einseitigen Hypothesentests ausgegeben:
> hund.bt.2sided <- binom.test(x = hund.kk.fa, n = kk.gr, p = 3.52e-05)
> hund.bt.2sided$p.value
[1] 0.06306491
> hund.bt.greater <- binom.test(x = hund.kk.fa, n = kk.gr, p = 3.52e-05, alternative="greater")
> hund.bt.greater$p.value
[1] 0.0321884
> hund.pt1.2sided <- prop.test(x = hund.kk.fa, n = kk.gr, p = 3.52e-05)
> hund.pt1.2sided$p.value
[1] 0.06349621
> hund.pt1.greater <- prop.test(x = hund.kk.fa, n = kk.gr, p = 3.52e-05, alternative="greater")
> hund.pt1.greater$p.value
[1] 0.0317481
Mit einem Zwei-Stichprobentest kann man überprüfen, ob ein Unterschied zwischen den Stichproben bezüglich des Werts einer Variable statistisch Signifikant ist, und damit, ob die zwei Stichproben aus derselben Grundgesamtheit stammen (in Bezug auf das gemessene Merkmal). Als Beispiel betrachten wir wieder die Häufigkeit des Wortes Hund in den zwei DWDS-Korpora Kernkorpus und Berliner Zeitung als Stichproben. Wie beim Ein-Stichprobentest zeigen wir die Berechnungen der Teststatistik (die beim Zwei-Stichprobentest wesentlich komplizierter ist als beim Ein-Stichprobentest) und des p-Werts im Detail als Hintergrundwissen. Anschließend sehen wie, wie dieser Test einfach und direkt in R durchgeführt wird.
H0: p(Hundkk) = p(Hundbz)
H1: p(Hundkk) ≠ p(Hundbz)
> kk.gr <- 121397601
> hund.kk.fa <- 4395
> bz.gr <- 237093180
> hund.bz.fa <- 7361
Für die Bestimmung der binomialverteilten Stichprobenverteilung gemäß der Nullhypothese müssen wir einen Schätzwert für den Parameter p der Auftretenswahrscheinlichkeit in der Grundgesamtheit verwenden. Die Nullhypothese erlaubt uns, die Summe der Stichprobengrößen und die Summe der ermittelten Häufigkeiten als Werte einer größeren Stichprobe aus derselben Grundgesamtheit und die entsprechende relative Häufigkeit als die geschätzte Auftretenswahrscheinlichkeit zu betrachten:
> (hund.kk.fa + hund.bz.fa) / (kk.gr + bz.gr) # =
(4395 + 7361)/(121397601 + 237093180) = 11756/358490781
[1] 3.279303e-05
Somit ergibt sich die Zufallsvariable:
X ∼ B(358490781, 3.279303e-05)binom.test() und ist folglich für typische
korpuslinguistische Stichproben nur bedingt anwendbar; daher verwenden
wir sie in diesem Seminar nicht. Aber wie beim Ein-Stichprobentest
gibt es für den Zwei-Stichprobentest eine nicht-exakte Alternative,
der
die Chi-Quadrat-Verteilung
zugrundeliegt. Für diese Alternative gibt es in R auch zwei
Funktionen: die schon beim Ein-Stichprobentest vorgestellte
Funktion prop.test() und die
Funktion chisq.test(). Beide implementieren den
in der Statistik berühmten Chi-Quadrat-Test für Kontingenztafeln. Da
diese viele Anwendungen in der Statistik haben, ist es sinnvoll als
Hintergrundwissen, die Berechnung des p-Werts nach dieser Methode
etwas näher zu betrachten. Diese beruht auf folgenden Überlegungen:
Um die erwarteten Häufigkeiten fe manuell zu berechnen, ist es praktisch, die ermittelten Werte der Stichproben in Form einer sogenannten 2×2-Kontingenztafel (auch Vierfelder-Tafel genannt) aufzustellen, die nicht nur die „Erfolge“ (in unserem Beispiel also die zwei beobachteten Häufigkeiten von Hund) sondern auch die „Misserfolge“ (die Häufigkeiten aller anderen Wortformen) enthält, damit alle Einheiten der Stichprobe berücksichtigt werden.
Die Kontingenztafel für unser Beispiel können wir in R mit Hilfe der
Funktion cbind() wie folgt erstellen (die Benennung der
Spalten und Zeilen durch die Funktionen colnames()
bzw. rownames() ist nicht notwendig aber verdeutlicht die
anschließenden Berechnungen):
> hund.kt <- cbind(c(hund.kk.fa, kk.gr - hund.kk.fa),
c(hund.bz.fa, bz.gr - hund.bz.fa))
> colnames(hund.kt) <- c("kk", "bz")
> rownames(hund.kt) <- c("Hund", "Nicht-Hund")
> hund.kt
kk bz
Hund 4395 7361
Nicht-Hund 121393206 237085819
Um die erwarteten Häufigkeiten zu berechnen, brauchen wir noch die Summen der Zeilen und Spalten:
> (hund.summe <- sum(hund.kt["Hund", ]))
[1] 11756
> (nicht.hund.summe <- sum(hund.kt["Nicht-Hund", ]))
[1] 358479025
> (kk.summe <- sum(hund.kt[ , "kk"])) # = kk.gr
[1] 121397601
> (bz.summe <- sum(hund.kt[ , "bz"])) # = bz.gr
[1] 237093180
> (kt.summe <- sum(hund.kt))
[1] 358490781
Diese Summen können wir mit der Funktion addmargins()
auch in der Kontingenztafel ausgeben:
> addmargins(hund.kt)
kk bz Sum
Hund 4395 7361 11756
Nicht-Hund 121393206 237085819 358479025
Sum 121397601 237093180 358490781
Die erwarteten Häufigkeiten ergeben sich aus dem jeweiligen Produkt der Summe einer Zeile und der Summe einer Spalte geteilt durch die Summe beider Zeilen (oder beider Spalten – die Summen sind ja gleich):
> (hund.kk.fe <- hund.summe * kk.summe / kt.summe) # 11756 * 121397601 / 358490781
[1] 3980.996
> (hund.bz.fe <- hund.summe * bz.summe / kt.summe) # 11756 * 237093180 / 358490781
[1] 7775.004
> (nicht.hund.kk.fe <- nicht.hund.summe * kk.summe / kt.summe) # 358479025 * 121397601 / 358490781
[1] 121393620
> (nicht.hund.bz.fe <- nicht.hund.summe * bz.summe / kt.summe) # 358479025 * 237093180 / 358490781
[1] 237085405
Jetzt können wir die Terme (fb − fe)2 / fe der Summe der quadrierten z-Werte berechnen:
> (hund.kk.z2 <- (hund.kk.fa - hund.kk.fe)^2 / hund.kk.fe)
[1] 43.05448
> (hund.bz.z2 <- (hund.bz.fa - hund.bz.fe)^2 / hund.bz.fe)
[1] 22.04496
> (nicht.hund.kk.z2 <- (hund.kt["Nicht-Hund", "kk"] - nicht.hund.kk.fe)^2 / nicht.hund.kk.fe)
[1] 0.001411933
> (nicht.hund.bz.z2 <- (hund.kt["Nicht-Hund", "bz"] - nicht.hund.bz.fe)^2 / nicht.hund.bz.fe)
[1] 0.000722945
Die Summe dieser quadrierten z-Werte ist der Wert der Chi-Quadrat-Teststatistik:
> (hund.z2.summe <- sum(hund.kk.z2, hund.bz.z2, nicht.hund.kk.z2, nicht.hund.bz.z2))
[1] 65.10158
Und zwar handelt es sich um einen Wert aus einer Chi-Quadrat-Verteilung mit einem Freiheitsgrad. Das ist so, weil die z-Werte nicht unabhängig voneinander sind: Da die Summen der Zeilen und der Spalten festgelegt sind, werden durch Festlegung eines der Werte der Kontingenztafel alle anderen Werte auch bestimmt. Daher ist nur einer der Werte frei variierbar.
Der gesuchte p-Wert ist also die Wahrscheinlichkeit
P(X2 ≥ 65,10158),
wo X2 ∼
χ2(1) eine Chi-Quadrat-verteilte
Zufallsvariable mit einem Freiheitsgrad ist. Diese
Wahrscheinlichkeit können wir mit der
R-Funktion pchisq() berechnen, der Verteilungsfunktion
der Chi-Quadrat-Verteilung:
> pchisq(q = hund.z2.summe, df = 1, lower.tail = FALSE)
[1] 7.113496e-16
Eine solche mühsame Berechnung der Chi-Quadrat-Teststatistik und des
p-Werts – macht man in der Praxis nicht in R, sondern ruft einfach
die Funktion chisq.test() oder
wiederum prop.test() auf:
chisq.test() nimmt ein obligatorisches
Argument x, das Daten in Form einer Kontingenztafel enthält.
prop.test() kann auch eine Kontingenztafel als
obligatorisches Argument nehmen, oder alternativ wie beim
Ein-Stichprobentest die Argumente x, n, die hier aber
zweistellige Vektoren der beobachteten Häufigkeiten und der
Stichprobengrößen sind. (Das beim Ein-Stichprobentest obligatorische
Argument p entfällt beim Zwei-Stichprobentest unter der
Nullhypothese, dass die beobachteten Werte aus derselben
Grundgesamtheit stammen.)
chisq.test()
und prop.test() das optionale
Argument correct. Mit dem voreingestellten Wert TRUE
kann es bei kleinen Stichproben einen genaueren p-Wert ergeben, aber
bei großen Stichproben, wie in der Korpuslinguistik üblich, ist eine
solche Korrektur unnötig oder kann sogar irreführend sein (siehe den
Chi-Quadrat-Test mit den Korpora Kernkorpus 21 und Berliner Zeitung im
nächsten Abschnitt).
prop.test() – aber
nicht chisq.test() – nimmt das optionale
Argument alternative mit denselben Werten wie beim
Ein-Stichprobentest. Mit alternative = "greater"
oder alternative = "less" wird der p-Wert bezüglich der
Normalverteilung anstatt der Chi-Quadrat-Verteilung berechnet. Da die
Normalverteilung symmetrisch ist, ist der p-Wert eines einseitigen
Chi-Quadrat-Tests genau die Hälfte des p-Werts des entsprechenden
zweiseitigen Tests.
> chisq.test(hund.kt)
Pearson's Chi-squared test with Yates' continuity correction
data: hund.kt
X-squared = 64.944, df = 1, p-value = 7.704e-16
> prop.test(c(hund.kk.fa, hund.bz.fa), c(kk.gr, bz.gr))
2-sample test for equality of proportions with continuity correction
data: c(hund.kk.fa, hund.bz.fa) out of c(kk.gr, bz.gr)
X-squared = 64.944, df = 1, p-value = 7.704e-16
alternative hypothesis: two.sided
95 percent confidence interval:
3.866289e-06 6.446684e-06
sample estimates:
prop 1 prop 2
3.620335e-05 3.104687e-05
> chisq.test(hund.kt, correct=FALSE)
Pearson's Chi-squared test
data: hund.kt
X-squared = 65.102, df = 1, p-value = 7.113e-16
> prop.test(c(hund.kk.fa, hund.bz.fa), c(kk.gr, bz.gr), correct=FALSE)
2-sample test for equality of proportions without continuity
correction
data: c(hund.kk.fa, hund.bz.fa) out of c(kk.gr, bz.gr)
X-squared = 65.102, df = 1, p-value = 7.113e-16
alternative hypothesis: two.sided
95 percent confidence interval:
3.872516e-06 6.440456e-06
sample estimates:
prop 1 prop 2
3.620335e-05 3.104687e-05
Wie man sieht, berechnen chisq.test()
und prop.test() identische Chi-Quadrat-Statistiken und
p-Werte. Man merke auch, dass diese Werte nur
mit correct=FALSE diesselben sind wie bei der obigen
manuellen Berechnung. Und schließlich berechnet
nur prop.test() auch ein Konfidenzintervall für die
Differenz der Häufigkeiten der Stichproben.
Wie beim Ein-Stichprobentest kann man den Rückgabewert dieser Funktionen einer Variable zuweisen und mit dem $-Operator auf die Elemente des Rückgabewerts, z.B. den p-Wert, zugreifen:
> hund.x2.corrT <- chisq.test(hund.kt) # oder: prop.test(hund.kt)
> hund.pt2.corrT <- prop.test(c(hund.kk.fa, hund.bz.fa), c(kk.gr, bz.gr))
> hund.x2.corrF <- chisq.test(hund.kt, correct=FALSE) # oder: prop.test(hund.kt, correct=FALSE)
> hund.pt2.corrF <- prop.test(c(hund.kk.fa, hund.bz.fa), c(kk.gr, bz.gr), correct=FALSE)
> hund.x2.corrT$p.value
[1] 7.704037e-16
> hund.pt2.corrT$p.value
[1] 7.704037e-16
> hund.x2.corrF$p.value
[1] 7.113496e-16
> hund.pt2.corrF$p.value
[1] 7.113496e-16
Den Chi-Quadrat-Test kann man auch auf mehr als zwei Stichproben anwenden. Wir können z.B. unser obiges Beispiel durch entsprechende Daten aus dem DWDS-Kernkorpus 21 ergänzen:
> kk21.gr <- 15469000
> hund.kk21.fa <- 525
> hund3.kt <- cbind(hund.kt, c(hund.kk21.fa, kk21.gr - hund.kk21.fa))
> colnames(hund3.kt) <- c("kk", "bz", "kk21")
> addmargins(hund3.kt)
kk bz kk21 Sum
Hund 4395 7361 525 12281
Nicht-Hund 121393206 237085819 15468475 373947500
Sum 121397601 237093180 15469000 373959781
Jetzt führen wir den Chi-Quadrat-Test durch und geben mit Hilfe des
$-Operators nicht nur den p-Wert sondern auch einige andere Elemente des
Rückgabewerts aus (N.B.: das Argument correct wird nur bei
Kontingenztafeln mit zwei Spalten und zwei Zeilen berücksichtigt, spielt
also hier keine Rolle):
> hund3.x2 <- chisq.test(hund3.kt)
> hund3.x2$expected # erwartete Werte gemäß der Nullhypothese
kk bz kk21
Hund 3.986749e+03 7.786242e+03 5.080086e+02
Nicht-Hund 1.213936e+08 2.370854e+08 1.546849e+07
> hund3.x2$parameter # die Freiheitsgrade = (3-1)×(2-1)
df
2
> hund3.x2$statistic # der Wert der Teststatistik
X-squared
65.60048
> hund3.x2$p.value # der p-Wert
[1] 5.689009e-15
Die Ergebnisse sind denen des obigen Zwei-Stichprobentests sehr ähnlich: Zwar ist die Teststatistik hier geringfügig größer aber bei zwei Freiheitsgraden anstatt eines, wodurch der p-Wert hier auch etwas größer ist als beim Zwei-Stichprobentest, aber dennoch so klein, dass die Nullhypothese, dass (bezüglich der Auftretenswahrscheinlichkeit des Wortes Hund) alle Stichproben aus derselben Grundgesamtheit stammen, mit großem Vertrauen abgelehnt werden kann.
Allerdings gilt diese Schlussfolgerung nur für alle drei Stichproben
zusammengenommen. Oben haben wir festgestellt, dass dieselbe
Schlussfolgerung auch für den Vergleich des DWDS-Kernkorpus und des
Korpus der Berliner Zeitung gilt. Es bleiben aber noch die Vergleiche
dieser Korpora jeweils mit dem Kernkorpus 21. Hier sind die
entsprechenden Hypothesentests mit Ausgabe der p-Werte (wir verwenden
hier prop.test() mit zweistelligen Vektoren, um uns die
Erstellung von Kontingenztafeln zu ersparen; außerdem sieht man hier,
dass der $-Operator direkt auf den Aufruf der Funktion angewendet werden
kann):
# Vergleich Kernkorpus 21 und Berliner Zeitung:
> prop.test(c(hund.kk21.fa, hund.bz.fa), c(kk21.gr, bz.gr))$p.value
[1] 0.05132178
# Vergleich Kernkorpus und Kernkorpus 21:
> prop.test(c(hund.kk.fa, hund.kk21.fa), c(kk.gr, kk21.gr))$p.value
[1] 0.1686542
Hier ist die Ablehnung der Nullhypothese in beiden Fällen statistisch nicht gerechtfertigt, wobei beim Vergleich Kernkorpus 21 und Berliner Zeitung das 5%-Signifikanzniveau nur knapp überschritten wurde. Das ist allerdings mit der Korrektur für kleine Stichproben, was hier eigentlich nicht zutrifft; ohne diese Korrektur ist der p-Wert für den Test Kernkorpus 21 und Berliner Zeitung wieder statistisch signifikant, wenn auch nur knapp:
> prop.test(c(hund.kk21.fa, hund.bz.fa), c(kk21.gr, bz.gr), correct = FALSE)$p.value
[1] 0.04857983
> prop.test(c(hund.kk.fa, hund.kk21.fa), c(kk.gr, kk21.gr), correct = FALSE)$p.value
[1] 0.1617964
Wenn man die relativen Häufigkeiten direkt miteinander vergleicht
(z.B. mit Hilfe von freq.rel()), stellt man folgende
Rangordnung fest: Kernkorpus (≈36,2 pMW) > Kernkorpus 21
(≈33,9 pMW) > Berliner Zeitung (≈31,0 pMW). Dann
könnte man entsprechende einseitige Tests durchführen:
> prop.test(c(hund.kk21.fa, hund.bz.fa), c(kk21.gr, bz.gr), alternative = "greater")$p.value
[1] 0.02566089
> prop.test(c(hund.kk.fa, hund.kk21.fa), c(kk.gr, kk21.gr), alternative = "greater")$p.value
[1] 0.08432709
Während der p-Wert für den Test Kernkorpus und Kernkorpus 21 immer noch die Nullhypothese unterstützt, liegt der p-Wert für den Test Kernkorpus 21 und Berliner Zeitung nach dem 5%-Signifikanzniveau jetzt deutlich im Ablehnungsbereich. Dennoch ist der Unterschied zum extrem kleinen p-Wert für den Test Kernkorpus und Berliner Zeitung, sowie für den Test aller drei Stichproben, sehr auffällig.
In den vorangehenden Beispielen ging es um den Vergleich der relativen Häufigkeiten (d.h. bezüglich der Stichprobengröße) einer Variable in zwei oder mehr Stichproben. Aber man kann mit einem Chi-Quadrat-Test auch proportionale Häufigkeiten zwischen Stichproben vergleichen. Dabei geht es um den direkten Vergleich der Werte, die sich als Ausprägungen des Merkmals auschließen, z.B. Substantive mit mehr als einer Pluralform oder Verben mit mehr als einer Form des Partizips. In solchen Fällen spielt die Korpusgröße keine Rolle, sondern die Stichprobe besteht nur aus der Gesamtheit der einzelnen Variablenwerte.
Als Beispiel nehmen wir die Verteilung nach Textklasse der Häufigkeiten
der zwei Varianten des Partizips II von senden,
also gesendet und gesandt, im DWDS-Kernkorpus. Wir testen
also die Verteilung einer Variable mit zwei Werten in vier Stichproben
(den Textklassen). Die Daten können wir am einfachsten anhand von
Count-Anfragen mit dwds.data.frame() in R einlesen:
> (gesendet.tk.kk <- dwds.data.frame(Anfrage = "count(@gesendet) #by[textClass~s/:.*//]"))
Anzahl key1
1 31 Belletristik
2 36 Gebrauchsliteratur
3 46 Wissenschaft
4 68 Zeitung
> (gesandt.tk.kk <- dwds.data.frame(Anfrage = "count(@gesandt) #by[textClass~s/:.*//]"))
Anzahl key1
1 217 Belletristik
2 161 Gebrauchsliteratur
3 107 Wissenschaft
4 251 Zeitung
Für die Erstellung einer Kontingenztafel aus diesen Häufigkeitstabellen
verwenden wir nun nicht cbind()
sondern rbind(), um die Anzahl-Spalten der Tabellen in
Zeilen der Kontingenztafel umzuwandeln:
> senden.p2.kt <- rbind(gesendet.tk.kk$Anzahl, gesandt.tk.kk$Anzahl)
> colnames(senden.p2.kt) <- gesendet.tk.kk$key1
> rownames(senden.p2.kt) <- c("gesendet", "gesandt")
> addmargins(senden.p2.kt)
Belletristik Gebrauchsliteratur Wissenschaft Zeitung Sum
gesendet 31 36 46 68 181
gesandt 217 161 107 251 736
Sum 248 197 153 319 917
Jetzt können wir chisq.test() durchführen:
> chisq.test(senden.p2.kt)$p.value
[1] 0.0002404428
Auch in diesem Beispiel ist der p-Wert des Hypothesentests deutlich kleiner als das traditionelle Signifikanzniveau von 5% – wenn auch bei weitem nicht so klein wie im vorangehenden Beispiel, was wohl auch mit der viel kleineren Stichprobe in diesem Fall zusammenhängt – und demnach wäre Nullhypothese, dass diese Belege von gesendet und gesandt aus derselben Grundgesamtheit stammen, abzulehnen.
Allerdings gilt wie im Beispiel im vorangehenden Abschnitt mit drei
Stichproben auch hier, dass eventuelle Unterschiede bezüglich
statistischer Signifikanz zwischen den Textklassen nur durch
Zwei-Stichprobentests ermittelt werden können. Dazu können wir mit
Hilfe von Indizierung aus dem
2×4-Kontingenztafel senden.p2.kt für jedes Paar von
Textklassen eine 2×2-Kontingenztafel erstellen und auf
diese chisq.test() jeweils anwenden und den p-Wert
ausgeben. Dabei verwenden wir jeweils das
Argument correct=FALSE, weil die Stichproben hier zwar
kleiner als die Stichproben in den Beispielen mit relativen Häufigkeiten
sind, gelten aber eigentlich nicht als klein im Sinne des Arguments:
> chisq.test(senden.p2.kt[ , c("Belletristik", "Gebrauchsliteratur")], correct = FALSE)$p.value
[1] 0.09069019
> chisq.test(senden.p2.kt[ , c("Belletristik", "Wissenschaft")], correct = FALSE)$p.value
[1] 1.438285e-05
> chisq.test(senden.p2.kt[ , c("Belletristik", "Zeitung")], correct = FALSE)$p.value
[1] 0.006082423
> chisq.test(senden.p2.kt[ , c("Gebrauchsliteratur", "Wissenschaft")], correct = FALSE)$p.value
[1] 0.009782062
> chisq.test(senden.p2.kt[ , c("Gebrauchsliteratur", "Zeitung")], correct = FALSE)$p.value
[1] 0.4025996
> chisq.test(senden.p2.kt[ , c("Wissenschaft", "Zeitung")], correct = FALSE)$p.value
[1] 0.03765723
Vier der sechs Tests zeigen ein statistisch signifikantes Ergebnis gemäß dem 5%-Signifikanzniveau. Nur beim Test Belletristik-Wissenschaft ist der p-Wert sehr klein und die Ablehnung der Nullhypothese wohl zweifelsfrei berechtigt. Auffällig ist, dass nach diesen Ergebnissen die Textklassen Belletristik und Gebrauchsliteratur zumindest tendenziell und die Textklassen Gebrauchsliteratur und Zeitung mit großer Wahrscheinlichkeit jeweils zur selben Grundgesamtheit (bezüglich der getesteten Wortformen) gehören, während die Textklassen Belletristik und Zeitung wohl zu unterschiedlichen Grundgesamtheiten gehören.
Wir können schließlich auch Zwei-Stichprobentests für den Vergleich der
Verteilung der Wortformen zwischen den einzelnen Textklassen und dem
Gesamtkorpus durchführen. Hierzu ist es nützlich, die
Kontingenztafel senden.p2.kt um die Randsummen zu ergänzen,
von Letzteren brauchen wir allerdings nur die Spalte der Summen, weil
diese die Gesamthäufigkeit jeder Wortform enthält (während die Zeile der
Summen die Gesamthäufigkeit beider Wortformen pro Textklasse enthält,
ist also für diesen Vergleich nicht geeignet):
> senden.p2.kt2 <- addmargins(senden.p2.kt)
> chisq.test(senden.p2.kt2[1:2 , c("Belletristik", "Sum")], correct = FALSE)$p.value
[1] 0.008762995
> chisq.test(senden.p2.kt2[1:2 , c("Gebrauchsliteratur", "Sum")], correct = FALSE)$p.value
[1] 0.6377929
> chisq.test(senden.p2.kt2[1:2 , c("Wissenschaft", "Sum")], correct = FALSE)$p.value
[1] 0.003821905
> chisq.test(senden.p2.kt2[1:2 , c("Zeitung", "Sum")], correct = FALSE)$p.value
[1] 0.544924
Hier gibt es eine ziemlich deutliche Trennlinie zwischen den Textklassen Gebrauchsliteratur und Zeitung auf der einen Seite, die nach diesen Ergebnissen zur selben Grundgesamtheit wie das Gesamtkorpus gehören (bezüglich der getesteten Wortformen), und auf der anderen Seite die Textklassen Belletristik und Wissenschaft, die wohl eher aus anderen Grundgesamtheiten als das Gesamtkorpus stammen (bezüglich der getesteten Wortformen).
Es ist nicht offensichtlich, wie man die Unterschiede in diesen Ergebnissen erklären soll, insbesondere, welche Einflüsse die verschiedenen Textklassen auf die Auftretenswahrscheinlichkeit unterschiedlicher Wörter haben. Weitere korpuslinguistische (aber auch z.B. lexikographische und grammatische) Recherchen, auch mit anderen Wörtern, könnten Faktoren aufdecken, die für solche Unterschiede verantwortlich sind oder mindestens mit ihnen statistisch zusammenhängen. Wenn weitere Hypothesentests mit vielen Wörtern auch solche unterschiedliche Ergebnisse zeigen und man keine naheliegenden Faktoren dafür findet, könnte das ein Indiz sein, dass die Stichproben die notwendigen statistischen Voraussetzungen, insbesondere, dass es sich um Zufallsstichproben handelt, nicht erfüllen, mit anderen Worten: dass die Korpora nicht repräsentativ für die Sprachdomäne sind.