Wahrscheinlichkeitsverteilungen mit R
Wenn eine Wahrscheinlichkeitsverteilung durch eine mathematische Formel
beschrieben werden kann, kann man diese Formel oft direkt in R
übersetzen. Nehmen wir als Beispiel die Wahrscheinlichkeitsfunktion
der Binomialverteilung
und ein Zufallsexperiment bestehend aus 10 Würfen einer fairen Münze,
d.h. mit der binomialverteilten Zufallsvariable X ∼ B(10,
0.5). Was ist die Wahrscheinlichkeit von 3 Köpfen bei den 10 Würfen? R
hat eine eingebaute Funktion für Fakultäten, factorial(),
die die Berechnung von Binomialkoeffizienten erleichtert. Damit können
wir mit folgenden Eingaben die Wahrscheinlichkeit berechnen:
-
> x <- 3
> n <- 10
> p <- 0.5
> factorial(n)/(factorial(x)*factorial(n-x)) * p^x * (1-p)^(n-x)
[1] 0.1171875
Die Wahrscheinlichkeit liegt also bei knapp 12%. Die Berechnung geht
aber einfacher, denn R hat auch eine eingebaute Funktion für den
Binomialkoeffizienten, choose():
-
> choose(n, x) * p^x * (1-p)^(n-x)
[1] 0.1171875
Und es geht sogar noch einfacher, denn R hat eine eingebaute Funktion
eigens für die Berechnung von binomialverteilten
Wahrscheinlichkeiten, dbinom():
-
> dbinom(x, n, p)
[1] 0.1171875
R-Funktionen für Wahrscheinlichkeitsverteilungen
Nicht nur für die Binomialverteilung sondern für viele bekannte
Wahrscheinlichkeitsverteilungen gibt es eingebaute R-Funktionen, und
zwar jeweils vier: für die Wahrscheinlichkeits(dichte)funktion (die
Zähldichte bei diskreten Zufallsvariablen bzw. die
Wahrscheinlichkeitsdichte bei stetigen Zufallsvariablen), für die
Verteilungs- und die Quantilfunktion, und schließlich für die Simulation
von Zufallsexperimenten mit Zufallswerten. Diese R-Funktionen beginnen
jeweils mit den Buchstaben ‚d‘, ‚p‘,
‚q‘ bzw. ‚r‘ (für random, also zufällig),
und nehmen zum Teil dieselben Argumente, zum Teil unterschiedliche, je
nach den Parametern der entsprechenden Wahrscheinlichkeitsverteilungen.
Hier ist zusammengefasst eine Beschreibung der R-Funktionen für die drei
vorhin vorgestellten Wahrscheinlichkeitsverteilungen:
- Binomialverteilung:
-
dbinom(x, size, prob)
pbinom(q, size, prob, lower.tail = TRUE)
qbinom(p, size, prob, lower.tail = TRUE)
rbinom(n, size, prob)
- Die Argumente
size und prob
entsprechen den Parametern n (Anzahl der Stufen des
Zufallsexperiments) bzw. p (Wahrscheinlichkeit des Erfolgs)
der Binomialverteilung.
- Normalverteilung:
-
dnorm(x, mean = 0, sd = 1)
pnorm(q, mean = 0, sd = 1, lower.tail = TRUE)
qnorm(p, mean = 0, sd = 1, lower.tail = TRUE)
rnorm(n, mean = 0, sd = 1)
- Die optionalen Argumente
mean und sd
nehmen den Erwartungswert bzw. die Standardabweichung der
Verteilung; falls diese Argumente weggelassen werden, werden die
Werte 0 bzw. 1 übergeben, d.h. die Verteilung ist dann die
Standardnormalverteilung.
- Chi-Quadrat-Verteilung:
-
dchisq(x, df)
pchisq(q, df, lower.tail = TRUE)
qchisq(p, df, lower.tail = TRUE)
rchisq(n, df)
- Das Argument
df nimmt die Anzahl der Freiheitsgrade
der Verteilung (df steht für die englische
Bezeichnung „degrees of freedom“).
- Erläuterung der gemeinsamen Argumente dieser Funktionen:
- Das Argument
x der d-Funktionen nimmt einen Vektor
von Zahlen: bei diskreten Verteilungen ganze Zahlen und bei
stetigen Verteilungen reelle Zahlen. Für jede Zahl
xi berechnet die Funktion den entsprechenden
Wert der Wahrscheinlichkeitsfunktion (also P(X =
xi)) bzw. der entsprechende Wert der
Wahrscheinlichkeitsdichte und gibt einen Vektor der Ergebnisse
zurück.
- Insbesondere steht bei
dbinom() jede Zahl
xi für die Anzahl der „Erfolge“
in einem Zufallsexperiment und der Rückgabewert ist die
entsprechende Wahrscheinlichkeit.
- Das Argument
q der p-Funktionen nimmt einen Vektor
von Werten (in diesem Zusammenhang heißen die Werte
Quantile) xi aus der Verteilung und die Funktion
berechnet die entsprechenden Wahrscheinlichkeiten gemäß dem Wert
von lower.tail und gibt einen Vektor der Ergebnisse
zurück.
- Das Argument
p der q-Funktionen nimmt einen Vektor
von Wahrscheinlichkeiten pi und die Funktion
berechnet die entsprechenden Quantile gemäß dem Wert
von lower.tail und gibt einen Vektor der Ergebnisse
zurück.
- Das optionale Argument
lower.tail der p- und
q-Funktionen bestimmt, welche Wahrscheinlichkeiten in die
Berechnung einbezogen werden: Mit dem voreingestellten
Wert TRUE oder bei Weglassung des Arguments sind das
P(X ≤ xi) (also Wahrscheinlichkeiten am
unteren Ende der Verteilung), und mit dem Wert FALSE
sind das P(X > xi) (also Wahrscheinlichkeiten
am oberen Ende der Verteilung).
- Das Argument
n der r-Funktionen gibt die Anzahl der
simulierten Zufallswerte entsprechend der verwendeten Verteilung
an. Die Funktion erzeugt diese Zufallswerte und gibt sie in einem
Vektor zurück.
Es gibt bestimmte Gesetzmäßigkeiten zwischen den d-, p- und
q-Funktionen:
- Bei diskreten Zufallsvariablen ist der Wert der p-Funktion für ein
Quantil n gleich der Summe der Werte der d-Funktion
bis n oder nach n (je nach dem Wert
von
lower.tail), d.h: pbinom(x, size=n,
prob=p) ist gleich sum(dbinom(0:x, size=n,
prob=p)) und pbinom(x, size=n, prob=p,
lower.tail=FALSE) ist gleich sum(dbinom((x+1):n, size=n,
prob=p)). Z.B.:
> pbinom(3, size=10, prob=0.5)
[1] 0.171875
> sum(dbinom(0:3, size=10, prob=0.5))
[1] 0.171875
> pbinom(3, size=10, prob=0.5, lower.tail=FALSE)
[1] 0.828125
> sum(dbinom(4:10, size=10, prob=0.5))
[1] 0.828125
- Bei stetigen Zufallsvariablen entspricht der Wert der p-Funktion für
ein Quantil n der Fläche des Graphen der
Wahrscheinlichkeitsdichte für das Intervall bis n; dieser Wert
lässt sich auch mit der d-Funktion mit Hilfe der
Funktion
integrate() annäherungsweise berechnen (n.b.: in
den folgenden Eingaben stehen ‚-Inf‘ und ‚Inf‘
für alle Werte kleiner bzw. größer als der Wert des anderen Arguments
– hier 1.96 – im Prinzip bis ins (negative bzw. positive)
Unendliche, und weil es sich um eine Approximation handelt, wird der
maximale Betrag des Fehlers mit ausgegeben):
> pnorm(1.96) # Standardnormalverteilung: μ = 0, σ = 1; nur Werte am unteren Ende der Verteilung
[1] 0.9750021
> integrate(dnorm, -Inf, 1.96)
0.9750021 with absolute error < 1.3e-06
> pnorm(1.96, lower.tail=FALSE) # nur Werte am oberen Ende der Verteilung
[1] 0.0249979
> integrate(dnorm, 1.96, Inf)
0.0249979 with absolute error < 1.9e-05
- Die Gesamtwahrscheinlichkeit ist immer 1:
> pbinom(3, size=10, prob=0.5) + pbinom(3, size=10, prob=0.5, lower.tail=FALSE)
[1] 1
> sum(dbinom(0:3, size=10, prob=0.5)) + sum(dbinom(4:10, size=10, prob=0.5))
[1] 1
> sum(dbinom(0:10, 10, 0.5))
[1] 1
> pnorm(1.96) + pnorm(1.96, lower.tail=FALSE)
[1] 1
> integrate(dnorm, -Inf, Inf)
1 with absolute error < 9.4e-05
- Die p- und q-Funktionen sind die Umkehrfunktionen voneinander:
> qbinom(pbinom(3, 10, 0.5), 10, 0.5)
[1] 3
> pbinom(qbinom(0.171875, 10, 0.5), 10, 0.5) # 0.171875 = pbinom(3, 10, 0.5)
[1] 0.171875
> qnorm(pnorm(1.96))
[1] 1.96
> pnorm(qnorm(0.9750021)) # 0.9750021 = pnorm(1.96)
[1] 0.9750021
Wie oben angemerkt berechnen die
Funktionen dnorm(), pnorm()
und qnorm() mit den voreingestellten Werten der
Argumente mean und sd Werte für die
Standardnormalverteilung. Folglich beinhalten diese Funktionen auch
die Standardisierung
(z-Transformation). Wir haben vorhin am Beispiel der
Normalverteilung X~N(16.5,3.245) darauf hingewiesen, dass man
durch Standardisierung die Wahrscheinlichkeit eines Wertes ≥ 20 in
einer Tabelle von z-Werte nachschlagen kann, und zwar mit dem
z-Wert (20 − 16,5)/3,245 ≈ 1,08. In R bekommt man
die Wahrscheinlichkeit einfach mit pnorm():
-
> pnorm(1.08, lower.tail = FALSE)
[1] 0.1400711
Da der z-Wert 1,08 abgerundet ist, ist auch die berechnete
Wahrscheinlichkeit entsprechend ungenau. Ein genaueres Ergebnis erhält
man, wenn man auch die Berechnung des z-Werts R überlässt:
-
> pnorm((20-16.5)/3.245, lower.tail = FALSE)
[1] 0.140387
Die Werte 16,5 und 3,245 sind aber der Erwartungswert bzw. die
Standardabweichung dieser Binomialverteilung, daher können wir diese
Werte den entsprechenden Argumenten von pnorm() übergeben,
ohne dabei den Wert 20 aus der Verteilung zu standardisieren:
-
> pnorm(20, mean = 16.5, sd = 3.245, lower.tail = FALSE)
[1] 0.140387
Stichprobenverteilung von Korpushäufigkeiten
Stichprobenverteilungen, denen eine Wahrscheinlichkeitsverteilung
zugrundeliegt, für die es in R entsprechende Funktionen gibt, kann man
mit Hilfe der passenden d-Funktionen erstellen und auch grafisch
darstellen. Ein Standardbeispiel in der Korpuslinguistik ist die
Verteilung der Korpushäufigkeit von Wortformen, von denen man annimt,
dass sie zwei Bedingungen erfüllt:
- Erstens: Die Wörter in der Grundgesamtheit (einer Sprachdomäne
oder einer Sprache im Allgemeinen) haben jeweils feste
Auftretenswahrscheinlichkeiten.
- Zweitens: Das Auftreten eines Wortes in einem repräsentativen Korpus
wird allein durch seine Auftretenswahrscheinlichkeit in der
Grundgesamtheit bestimmt, also unabhängig vom Auftreten anderer Wörter
im Korpus. Folglich gilt bei der Durchführung einer Anfrage nach
einem bestimmten Wort im Korpus: Entweder gibt es Treffer
(„Erfolg“) oder es gibt keine („Misserfolg“),
und die Wahrscheinlichkeit des Erfolgs ist gleich der
Auftretenswahrscheinlichkeit in der Grundgesamtheit. Mit anderen
Worten: Die Verteilungen der Korpushäufigkeiten von Wortformen sind
binomialverteilte Zufallsvariablen.
Diese Annahmen stellen zugegebenermaßen Vereinfachungen der
Wirklichkeit dar, aber solche Vereinfachungen gehören zur üblichen
wissenschaftlichen Vorgehensweise beim Versuch, die Wirklichkeit durch
mathematische Modelle abzubilden. Die zweite Annahme insbesondere
trifft im normalen Sprachgebrauch, wo der Kontext das Auftreten von
Wörtern sehr wohl beeinflusst, im Allgemeinen natürlich nicht zu (zumal
auf der Satzebene), dennoch: Die „Rechtfertigung für die
Binomial-Annahme basiert auf der grundsätzlich plausiblen Vermutung,
dass die relevanten Einflüsse auf die Auftretenswahrscheinlichkeit eines
Wortes relativ lokal sind (also nicht über große Entfernungen im Korpus
wirken) und sich über eine große Zahl von Texten gegenseitig
neutralisieren, so dass bei einem sehr großen Korpus die Gesamtfrequenz
des Wortes tatsächlich fast ausschließlich von seiner (angenommenen)
festen Auftretenswahrscheinlichkeit beeinflusst wird“
(Perkuhn et al., S. 89).
Als Beispiel erstellen wir die Stichprobenverteilung des
Wortes Hund im DWDS-Kernkorpus. Zunächst ermitteln wir die
absolute Häufigkeit anhand des Suchbegriffs ‚@Hund‘ und die
relative Häufigkeit bezüglich der Korpusgröße wie gehabt mit Hilfe der
Funktion freq.rel() (die ggf. mit
dem source()-Aufruf
in R geladen werden muss). Für Berechnungen in R mit diesen Werten
sollen sie derselben Größenordnung sein, daher lassen wir die relative
Häufigkeit als Dezimalzahl ausgeben (und der Anschaulichkeit wegen auch
in pMW):
-
> hund.kk.fa <- 4395
> kk.gr <- 121397601
> (hund.kk.fr <- freq.rel(hund.kk.fa, kk.gr, "dezimal"))
[1] 3.620335e-05
> (hund.kk.pmw <- freq.rel(hund.kk.fa, kk.gr))
[1] 36.20335
Für die Erstellung der Verteilung können wir natürlich nicht alle
möglichen Stichproben aus der Grundgesamtheit berücksichtigen, aber für
unsere Zwecke ist eine Stufigkeit von einer Million eine ausreichende
und auch sinnvolle Annäherung, weil wir die relative Häufigkeit
normalerweise auch in pMW berechnen. Demnach handelt es sich um die
Stichprobenverteilung der binomialverteilten Zufallsvariable X
∼ B(1e6, 3.620335e-05).
Für die grafische Darstellung dieser Verteilung könnten wir im Prinzip
alle der einen Million Wahrscheinlichkeiten, die sich aus der gewählten
Stufigkeit ergeben, berechnen und grafisch abbilden, aber ganz abgesehen
vom Rechenaufwand für den Computer ist es auch gar nicht sinnvoll so
viele Werte zu verwenden, denn aufgrund der Wahrscheinlichkeitsfunktion
der Binomialverteilung können wir feststellen, dass die allermeisten
dieser Wahrscheinlichkeiten verschwindend klein und daher erst recht
grafisch vernachlässigbar sind. Mit Hilfe der
Funktion qbinom() können wir eine für die grafische
Darstellung ausreichende Anzahl der grafisch abzubildenden
Wahrscheinlichkeiten bestimmen, indem wir der Funktion eine sehr kleine
Wahrscheinlichkeit übergeben, z.B. eine Millionstel (1e-6 in der
Exponentialdarstellung):
-
> n <- 1e6
> p <- 1e-6
> qbinom(p, n, hund.kk.fr)
[1] 12
> qbinom(p, n, hund.kk.fr, lower.tail=FALSE)
[1] 68
Diese Ergebnisse bedeuten, dass in einer Stichprobe aus dieser
Grundgesamtheit (d.h. in einem dem DWDS-Kernkorpus vergleichbaren
Korpus) die Wahrscheinlichkeit, dass die relative Häufigkeit
von Hund entweder nicht mehr als 12 pMW oder nicht weniger als 68
pMW beträgt, bei jeweils mindestens 1e-6 oder 0,0001% liegt und
folglich, dass die Wahrscheinlichkeit, eine relative Häufigkeit
innerhalb dieser zwei Grenzbereiche bei einer Suche festzustellen, bei
2e-6 oder 0,0002% liegt.
- Tatsächlich ist die Wahrscheinlichkeit höher: 4.502651e-06 oder
rund 0,00045%. Das ergibt sich daraus, dass die Binomialverteilung
eine diskrete Verteilung ist, es gibt also Lücken zwischen den
Wahrscheinlichkeiten aufeinderfolgender Quantile:
> pbinom(12, n, hund.kk.fr)
[1] 2.94145e-06
> pbinom(11, n, hund.kk.fr)
[1] 9.387865e-07
> pbinom(67, n, hund.kk.fr, lower.tail=FALSE)
[1] 1.561201e-06
> pbinom(68, n, hund.kk.fr, lower.tail=FALSE)
[1] 8.072584e-07
Wir haben 1e-6 als die kleinste zu berücksichtigende
Wahrscheinlichkeit festgelegt und die Quantile 12 und 68
unterschreiten diese Grenze nicht, im Gegensatz zu 11 und 69, den
nächsten Quantilen nach unten bzw. nach oben (N.B.: wie oben erwähnt
berücksichtigt pbinom() mit lower.tail=FALSE
nur größere Werte als der angegebene Wert, für pbinom(67,
...) also 68 und größer und für pbinom(68, ...) 69
und größer).
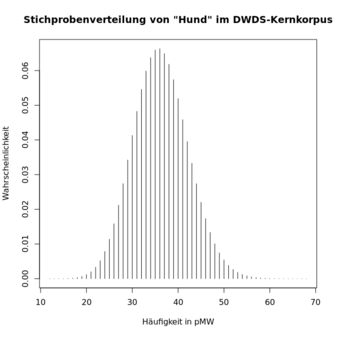
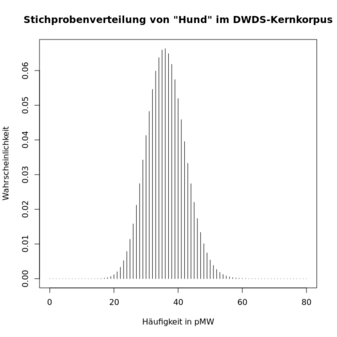
Wir können aber diese Quantile getrost auf 0 und 80 ab- bzw. aufrunden
und damit alle Werte am unteren Ende berücksichtigen (kleiner als 0
geht’s ja nicht). Die zwei sich ergebenden Grafiken (die ersten
zwei unten) ähneln sich sehr. Es ist auch klar zu erkennen, dass alle
Quantile größer als ca. 60 eine Wahrscheinlichkeit nahe Null haben.
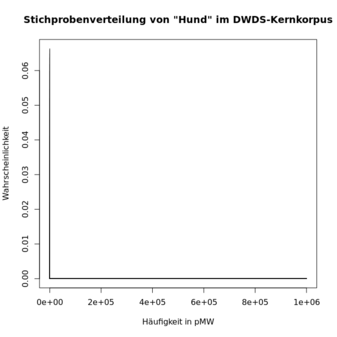
Daher zeigt eine grafischen Darstellung aller Quantile der gewählten
Stufigkeit von einer Million (die dritte Grafik unten), dass fast alle
Quantile und fast die gesamte Wahrscheinlichkeit in der Nähe von 0
liegen, was die eigentlich „interessanten“
Wahrscheinlichkeiten verdeckt:
-
> x <- 12:68 # wiederholen mit 0:80 bzw. 0:1e6 für die zweite bzw. dritte Grafik
> probs <- dbinom(x, n, hund.kk.fr)
> plot(x, probs, type="h", main="Stichprobenverteilung von \"Hund\" im DWDS-Kernkorpus", xlab="Häufigkeit in pMW", ylab="Wahrscheinlichkeit")
Die Erstellung der Stichprobenverteilung aus einem ermittelten Wert
(z.B. der Korpushäufigkeit) ist der Ausgangspunkt für Rückschlüsse von
der Stichprobe auf die zugrundeliegende Grundgesamtheit. Welche
Rückschlüsse gezogen werden können, hängt von der statistischen
Signifikanz des Werts, die man anhand von sogenannten Signifikanztests
überprüfen kann; darauf gehen wir im letzten Themenblock des Seminars im
Detail ein. An dieser Stelle sei nur noch einmal betont: Die Gültigkeit
der Tests und folglich auch der Rückschlüsse, setzt die Gültigkeit der
zugrundeliegenden Annahmen über die Stichprobenverteilung, wie die zwei
oben erwähnten für die Binomialverteilung, voraus.
Simulierte Zufallswerte
Es ist meistens zu aufwänding (mindestens zeitlich), viele Stichproben
aus einer Grundgesamtheit zu ziehen und Statistiken aus diesen mit den
erwarteten Werten gemäß der Stichprobenverteilung zu vergleichen. Aber
mit den r-Funktionen für die von R unterstützten
Wahrscheinlichkeitsverteilungen kann man solche wiederholte Stichproben
mühelos simulieren. Schon mit ca. 500 simulierten Zufallswerten kann
man die Gestalt der Stichprobenverteilung gut erkennen.
Als Beispiel erstellen wir Verteilungen von Zufallswerten für unsere
Zufallsvariable der Auftretenswahrscheinlichkeit von Hund im
DWDS-Kernkorpus. Das Ergebnis von rbinom() ist ein Vektor
von simulierten absoluten Häufigkeiten. Mit table()
erstellen wir die entsprechende Häufigkeitsverteilung; da wir aber nicht
die Verteilung der absoluten Häufigkeiten sondern wie bei der
Stichprobenverteilung die Verteilung der Auftretenswahrscheinlichkeiten
simulieren wollen, teilen wir die absoluten Häufigkeiten in der
Häufigkeitstabelle durch die gewählte Anzahl der Zufallswerte und
erhalten damit die relativen Häufigkeiten (die gemäß der zweiten
Bedingung oben die angenommenen Auftretenswahrscheinlichkeiten
sind):
-
> w <- 500
> hund.kk.zw <- rbinom(w, n, hund.kk.fr)
> hund.kk.zw.tab <- table(hund.kk.zw)/w
Für die grafische Darstellung dieser Verteilung müssen wir nur diese
Tabelle der plot()-Funktion übergeben. Durch das
Argument type="h" erscheinen die Wahrscheinlichkeiten als
dünne Säulen oder Stäbe ("h" steht für Histogramm, eine Art
Säulendiagramm). Für den direkten Vergleich mit der oben erstellten
Stichprobenverteilung wird mit dem Argument xlim=c(0,80)
nur Zufallswerte zwischen 0 und 80 gezeigt (andere sind ohnehin so
unwahrscheinlich, dass wir sie nicht berücksichtigen müssen). Damit die
Markierung der x-Achse übersichtlich wird, unterbinden wir zunächst mit
dem Argument xaxt="n" die automatische Markierung und
erstellen dann mit der Funktion axis() eigene Markierungen
(das Argument 1 bewirkt die Markierung der x-Achse (2 ist für die y-Achse)
und das Argument at ist ein Vektor der Markierungen, hier
in Zehnerschritten von 0 bis 80):
-
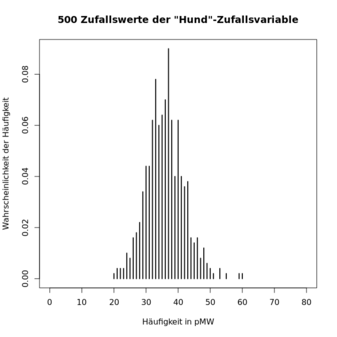
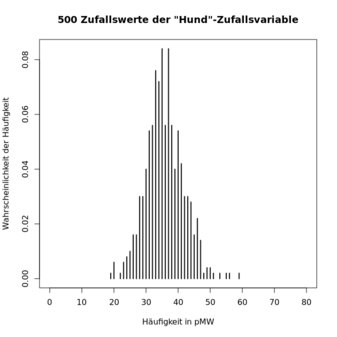
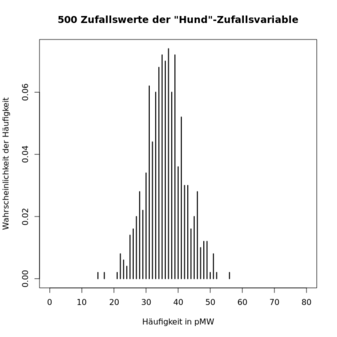
> plot(hund.kk.zw.tab, type="h", xlim=c(0,80), xaxt="n", main="500 Zufallswerte der \"Hund\"-Zufallsvariable", xlab="Häufigkeit in pMW", ylab="Wahrscheinlichkeit der Häufigkeit")
> axis(1, at=seq(0,80,by=10))
Mit jeder Wiederholung der letzten vier Eingaben wird eine neue
Zufallsverteilung dieser Zufallsvariable grafisch dargestellt. Hier
sind drei Beispiele, in denen die Ähnlichkeiten der zentralen Tendenz
und der Streuung der Verteilungen klar erkennbar sind, aber auch die
zufälligen Unterschiede, genau wie bei echten Zufallsstichproben:
Eine Folge des zentralen Grenzwertsatzes
Mit solchen Zufallsverteilungen kann man sehr schön eine Folge des
zentralen Grenzwertsatzes der Wahrscheinlichkeitstheorie nachvollziehen:
Wenn man in einem Zufallsexperiment eine Verteilung erstellt und daraus
eine Statistik, z.B. das arithmetische Mittel, berechnet und dasselbe
Experiment sehr oft wiederholt, ergibt sich eine Verteilung der
Statistik, die sich bei zunehmenden Wiederholungen immer mehr einer
Normalverteilung nähert (auch wenn die zugrundeliegende Verteilung keine
Normalverteilung ist). Hier ist ein Beispiel anhand
unserer Hund-Zufallsvariable:
- Wir erstellen eine Zufallsverteilung mit 500 Werten wie oben und
berechnen daraus das arithmetische Mittel. Wir wollen diesen Vorgang
aber viele Male wiederholen, was wir in R mit Hilfe einer
sogenannten for-Schleife (einer Programmiertechnik für
Wiederholungen) tun können. Mit jedem Durchlauf durch die Schleife
speichern wir den berechneten Wert in einem Vektor (der Vektor muss
zuerst durch Zuweisung initialisiert, d.h. R bekannt gemacht werden;
das R-Schlüsselwort
NULL dient als Platzhalter-Wert für
die Initialisierung des Vektors). Hier z.B. wird zehnmal eine
simulierte Verteilung erstellt und das entsprechende arithmetische
Mittel berechnet:
> rm <- NULL
> for (i in 1:10) rm[i] <- mean(rbinom(500, 1e6, hund.kk.fr))
- Eine einfache Möglichkeit zu überprüfen, ob sich die Verteilung der
Mittelwerte einer Normalverteilung nähert, ergibt sich aus der
Tatsache, dass bei einer Normalverteilung immer die gleichen Anteile
der Werte innerhalb von einer, zwei und drei Standardabweichungen vom
arithmetischen Mittel der Verteilungen liegen, und zwar: ca. 68,2%,
ca. 95,4% bzw. ca. 99,7% der Werte. Wir können die entsprechenden
Anteile für die Verteilung der Mittelwerte mit Hilfe von Indizierung
wie folgt berechnen (Sie können sich überzeugen, dass und warum dies
funktioniert):
# Mittelwert der Mittelwerte der 10 Stichproben:
> (rmm <- mean(rm))
[1] 36.0516
# Standardabweichung der Mittelwerte der 10 Stichproben:
> (rmsd <- sd(rm))
[1] 0.2721777
# Anteil der Mittelwerte innerhalb einer Standardabweichung:
> (length(rm[rm > rmm-rmsd & rm < rmm+rmsd])/10)
[1] 0.7
# Anteil der Mittelwerte innerhalb von zwei Standardabweichungen:
> (length(rm[rm > rmm-2*rmsd & rm < rmm+2*rmsd])/10)
[1] 1
# Anteil der Mittelwerte innerhalb von drei Standardabweichungen:
> (length(rm[rm > rmm-3*rmsd & rm < rmm+3*rmsd])/10)
[1] 1
Für diese Verteilung der Mittelwerte aus 10 Zufallsstichproben mit je
500 Werten liegen also 70% der Mittelwerte innerhalb von einer
Standardabweichung und schon 100% innerhalb von zwei (und folglich auch
innerhalb von drei) Standardabweichungen vom arithmetischen Mittel.
Diese Verteilung ist also nicht genau normal aber auch nicht völlig
anders. Aber es sind ja nur 10 Stichproben. Nach dem zentralen
Grenzwertsatz erwarten wir bei immer mehr Stichproben eine immer
genauere Annäherung an eine Normalverteilung.
Um solche Simulationen mit noch mehr Stichproben und auch mit
verschiedenen Suchergebnissen aus verschiedenen Korpora einfach
durchzuführen, haben wir eine R-Funktion definiert (die mit
unserem source()-Aufruf
in R geladen wird), am.zufall(). Diese Funktion nimmt drei
Argumente, die alle Zahlen als Werte haben.
- Das erste Argument,
freq.fa, nimmt als Wert die
absolute Häufigkeit des verwendeten Suchbegriffs, das zweite
Argument, korp.gr, nimmt als Wert die Größe des
verwendeten Korpus.
- Das dritte Argument,
n, gibt die Anzahl der
Simulationen (mit je 500 Werten) an, wobei sich mit jeder Wiederholung
der Simulation die Anzahl der Stichproben in der Simulation
verzehnfacht, d.h. n=1 bedeutet eine Simulation mit 10
Stichproben, n=2 bedeutet zwei Simulationen, eine mit 10
und eine mit 100 Stichproben, usw. bis maximal n=6, die
sechs Simulationen mit 10, 100, 1000, 10.000, 100.000 bzw. eine
Million Stichproben.
- Für jede Simulation wird eine grafische Darstellung der
Verteilung der Mittelwerte erstellt.
- Zusätzlich wird eine Tabelle in die R-Konsole ausgegeben, in der die
Spalten jeweils den Anteil der Mittelwerte innerhalb der ersten,
zweiten bzw. dritten Standardabweichung zeigen und die Zeilen die
Werte pro Simulation zeigen.
- Achtung: Mit
n=5 können die Berechnungen mehrere
Sekunden dauern und mit n=6 je nach Rechenleistung sogar
eine Minute oder länger.
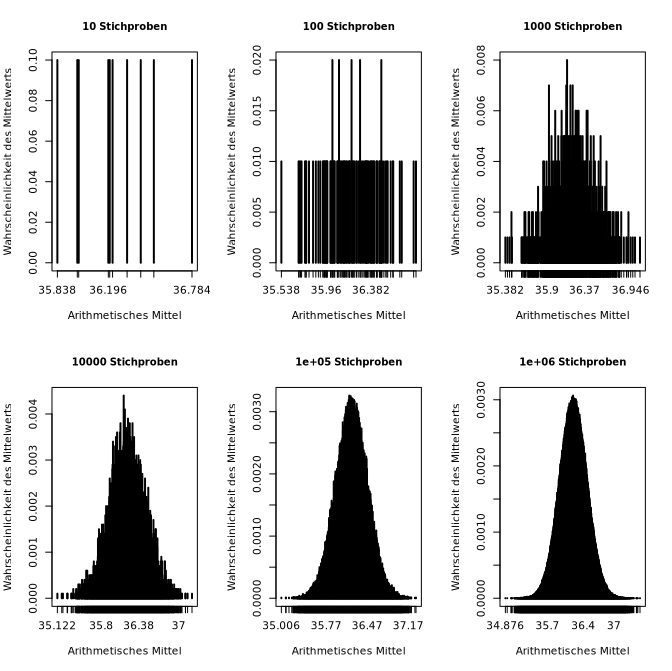
Mit folgendem Aufruf dieser Funktion werden sechs Simulationen für das
Wort Hund im DWDS-Kernkorpus durchgeführt; an den Grafiken und
den Zahlen ist die zunehmende Annäherung an eine Normalverteilung
deutlich und schon ab 1000 Wiederholungen erkennbar (n.b.: da es sich um
Zufallsverteilungen handelt, unterscheiden sich die Ergebnisse bei jeder
Durchführung, bei 100.000 und einer Million Stichproben allerdings nur
leicht):